超純水對於新世代基因體定序的影響
自從1976年Frederick Sanger等人提出DNA定序方法以來,經過長達36年的努力,最新一代的定序方法:氫離子半導體定序,已能夠使一次基因體定序的成本降至1000元美金,並且在8小時內完成;然而利用這種最新定序方法所得到的資料,其準確性和水質有很大的關聯性。
第一代定序:Sanger method
1976年Frederick Sanger等人提出雙脫氧鏈終止法 (dideoxy chain-termination method)作為DNA定序的方法,這種定序方法的原理是在反應時分別添加ddNTPs作為終止物 (chain terminator),並且經過PCR反應後,藉由電泳分析不同長度的DNA片段帶的終止物種類,得到序列資訊。這種方法經過修改,在1986年改成以不同螢光標記的ddNTPs作為終止物,並搭配自動化儀器的使用,使得這方法成為定序領域中的主流方法。
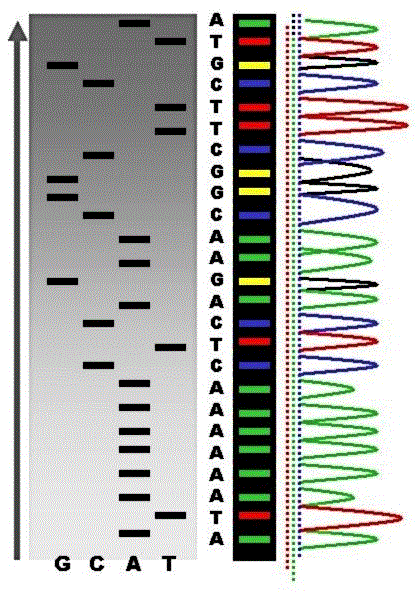
圖1. 將具有放射性標記的ddNTPs改以四種不同的螢光基團標記,並搭配毛細管電泳及自動化儀器,使得Sanger定序法成為DNA定序領域中最主流的方法。
次世代定序
Sanger sequencing操作流程簡單,而且一次反應得到的正確資料量約為1000 bp,非常適合小片段的DNA樣本分析。然而,將Sanger sequencing應用在基因體定序時,由於需要經過基因轉殖及質體放大的過程,因此一次定序所需的時間長 (約6-8周),而且最終的序列正確性還會受到轉殖序列偏差的影響 (Cloning bias)。
為了解決這些問題,乳化PCR技術 (Emulsion PCR)被使用在模版DNA分子製備階段中。這技術以乳化的方式將每一個模版DNA分子均勻分散至油滴中,每個油滴中含有一個球狀微粒,模版DNA分子此時利用微粒上的引子,油滴內的酵素和試劑,進行PCR放大反應。
偵測的過程也同時和PCR放大反應結合,依序加入不同的dNTPs時,只要序列和模版DNA相符,即能進行合成反應並且產生訊號。這些改進使得整個基因體定序過程中不須再經過轉殖和質體放大毛細管電泳,大大降低了一次定序所需時間及提高了正確率;這時期發展出的技術通稱為次世代定序 (Next generation sequencing,NGS),因為其高通量輸出的特點,使得反應成本和時間皆大幅降低。
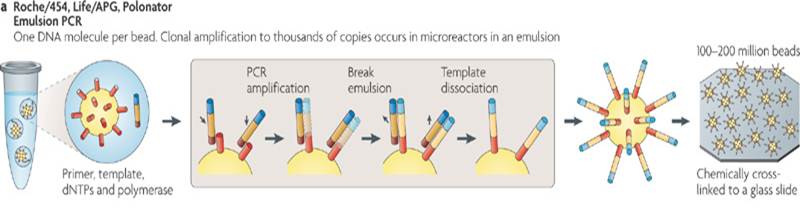
圖2. 經過Emulsion PCR (emPCR)技術處理,每一個模版分子經PCR放大後被固定在一個微粒子上,並且將這些微粒子以陣列的方式排列於反應版上,在定序的過程中提供高通量的定序資料輸出。
最新定序方法 - 氫離子半導體定序 (Hydorgen ion semiconductor sequencing)
在許多次世代定序技術中,仍然使用含有螢光團的的物質作為訊號來源,因此必須以光學儀器(通常為CCD照相機)進行偵測,才能正確地將化學反應中所提供的訊息轉換成數位訊號。基因體定序的成本也因為複雜的光學機構和螢光團標記物質無法降低。
終於在2011年,Jonathan M. Rothberg等人提出了革命性的技術,讓DNA定序從此擺脫光學的束縛,進入數位的時代。這項全新的技術利用氧化鉭,用以偵測反應槽內由DNA polymerase於反應的過程中放出的氫離子所造成的pH改變,接著由離子選擇場效電晶體(Ion Selective Field Effect Transistor,ISFET)進行電流訊號放大,即可讀取目標序列資訊。
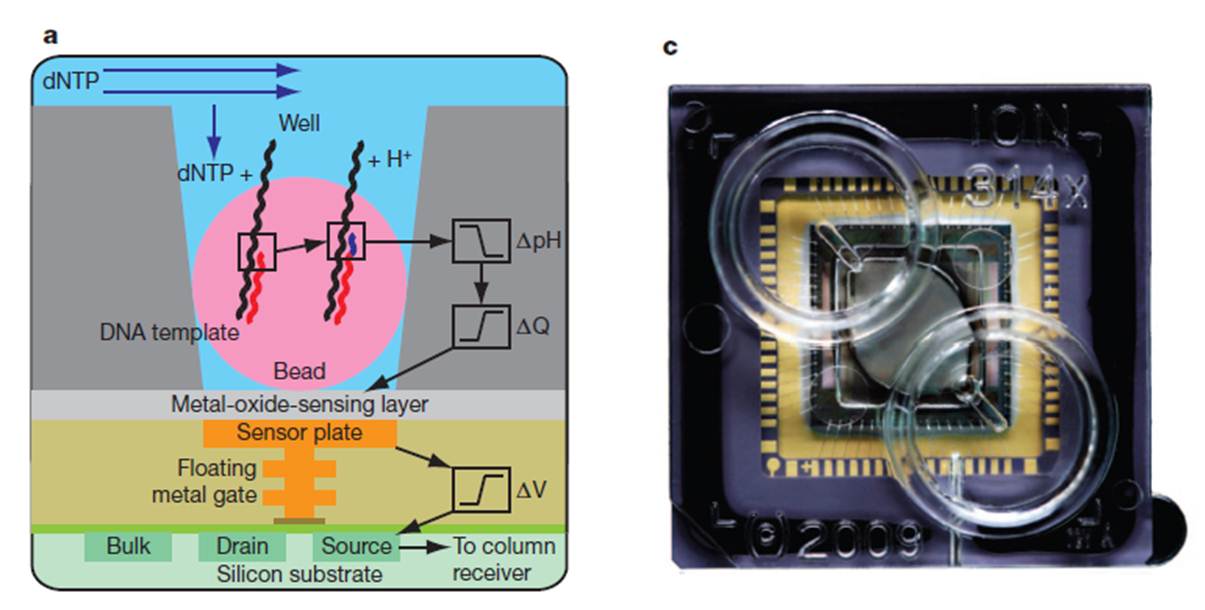
圖3 當加入符合目標序列的dNTPs時,氫離子會因DNA polymerase的合成作用而被放出,造成反應槽內pH的改變,此時會誘導氧化鉭層產生電位差的變化,造成下方的電晶體產生放大電流,完成化學和數位訊號間的轉換。
數位定序對於水質的要求
1. 低離子強度:氫離子的改變所造成的電流很微弱,僅為10-6安培而已,因此水中的任何鹽類污染都會造成假訊號的產生,使得定序結果產生錯誤。為了避免這種情況發生,反應用水的導電度應該要控制在0.055 µS/cm 以下 (即電阻率高於18.2 MW)。
2. 低有機物污染:氧化鉭層很容易附著有機污染物,一但被有機物污染,氧化鉭即會喪失其感受微小pH變化的敏感性,使得定序結果發生缺失。為了防止這種情況,反應用水的TOC值應該要控制在5 ppb以下。
3. 酵素污染:樣本中的DNA或RNA非常容易被微生物分泌的DNase和RNase分解,而使得原本copy number低的樣本無法被偵測,因此反應用水中不能有任何的DNase和
RNase污染。
參考資料
1. Nature Vol 437 p376-380, 15 September 2005, doi:10.1038/nature03959, Genome sequencing in microfabricated high-density picolitre reactors.
2. Nature Reviews Genetics Vol 11 p31-46, January 2010,doi:10.1038/nrg2626, Sequencing technologies — the next generation.
3. Nature Vol 475 p348-352, 21 July 2011, doi:10.1038/nature10242, An integrated semiconductor device enabling non-optical genome sequencing.
